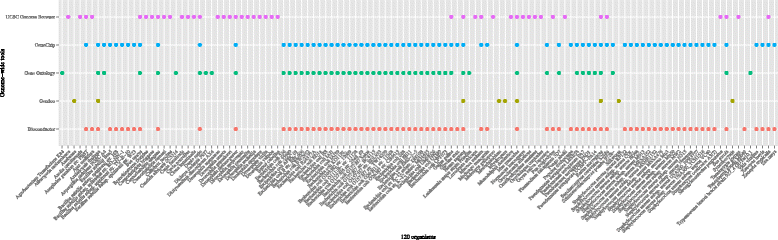
大規模1細胞データベース(DB)から、類似細胞を高速検索するソフトウェア「CellFishing.jl」を開発しました。
本研究成果は、細胞分化や臓器・器官発生などの基礎研究から、再生医療における移植細胞の有効性・安全性評価、創薬などの発展に貢献すると期待できます。
多細胞生物が持つ数百種類の細胞の機能を理解する方法として、1細胞ごとにRNAの種類と量を計測する「1細胞RNAシーケンス法]」があります。これにより、共同研究チームを含め、世界中が協力してヒトの全細胞種の遺伝子発現データをデータベース化する研究が進行中で、次々とデータが公開されています。世界中の研究者がこれらのDBと自分のデータを比較できれば、疾患の原因や薬剤応答を細胞レベルで精緻に理解できるようになります。
今回、我々は、1細胞発現データを検索するアルゴリズムを開発し、百万個の細胞DBから、高い検索精度を保ちつつも、1細胞あたり0.63ミリ秒で類似細胞の検索に成功しました。これは既存の方法の100倍以上の速さです。さらに、類似細胞で特徴的に働く遺伝子を高速に検出する機能も開発しました。
本研究は、英国の科学雑誌『Genome Biology』(2月11日付)に掲載されました。詳細は以下の日本語プレスリリースをご覧ください。
理研からのプレスリリース: 高速検索エンジン「CellFishing.jl」を開発 -大規模1細胞データベースから類似細胞を瞬時に検出する手法-
AMEDからのプレスリリース: 高速検索エンジン「CellFishing.jl」を開発 -大規模1細胞データベースから類似細胞を瞬時に検出する手法-
Kenta Sato, Koki Tsuyuzaki, Kentaro Shimizu, Itoshi Nikaido, “CellFishing.jl: an ultrafast and scalable cell search method for single-cell RNA-sequencing”, Genome Biology, 10.1186/s13059-019-1639-x