ABOUT US
国立研究開発法人 理化学研究所 生命機能科学研究センター バイオインフォマティクス研究開発チーム (Laboratory for Bioinformatics Research, BiT) のウェブサイトです。研究室のミッションは2つです。
- オミックスの新しいデータ解析手法、実験手法の研究開発
- バイオインフォマティクス、1細胞オミックス技術を通じた共同研究
研究チームについて詳しく知りたい方は About をご覧ください。研究活動や研究会などのお知らせはこちらです。人材募集についてはこちらをご覧ください。
SELECTED WORK
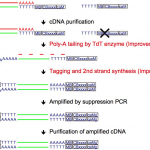 Quartz-Seq2: 低コスト、ハイスループットを兼ね備えた世界最高精度の1細胞RNA-seq法
Quartz-Seq2: 低コスト、ハイスループットを兼ね備えた世界最高精度の1細胞RNA-seq法
Quartz-Seq2は、1536種類の細胞バーコードを利用して、1細胞をプールして反応できるため、低コストに大量の1細胞RNA-seqが実施できます。また、RNAを効率的にシーケンスライブラリに変換することができるため、少ないシーケンスリードで多くの遺伝子を検出できます。この変換効率を示すUMI変換効率は32-52%あり、ほかの手法(7-22%)を大きく上回ります さらに、高効率のPolyA talingを用いたtagging、逆転写酵素量の削減、分子バーコードの採用などにより、低コスト化と高精度化を両立させました。
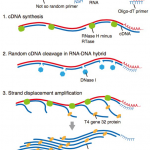 RamDA-seq: 完全長Total RNAシーケンスできる唯一の1細胞RNA-seq法
RamDA-seq: 完全長Total RNAシーケンスできる唯一の1細胞RNA-seq法
RNA逆転写時にcDNAを増幅できる新規核酸増幅法RT-RamDAとnot-so-random primingにより、遺伝子発現検出の超高感度化と、non-polyA RNAの検出、完全長からのcDNA合成を同時に達成しました。これにより、1細胞で non-polyA, pre-mRNA, enhanser RNAの検出に成功しました。通常の1細胞RNA-seqと比較しても、検出遺伝子数、遺伝子全長のカバー率が高く、実験間のばらつきが低いことが示されています。
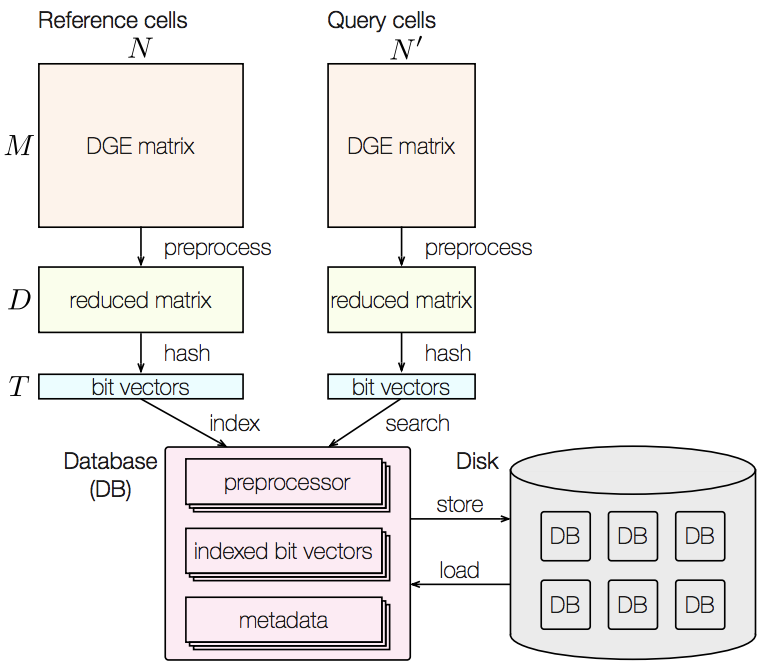 CellFishing.jl: 高速で正確な細胞検索ソフトウェア
CellFishing.jl: 高速で正確な細胞検索ソフトウェア
巨大な1細胞RNA-seqデータベースに対して、自分で計測した1細胞RNA-seqのデータを1細胞ずつ検索するソフトウェアCellFishing.jlを開発しました。このソフトウェアでは、Randomized singular value decompositionとlocality sensitive hashingを組み合せた最近傍検索を用いています。データのインデックス化やデータ構造も工夫して、高速に正確に細胞検索ができるようになりました。
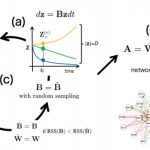 SCODE: 時系列1細胞RNA-seqデータからの効率のよい転写制御ネットワーク予測アルゴリズム
SCODE: 時系列1細胞RNA-seqデータからの効率のよい転写制御ネットワーク予測アルゴリズム
このアルゴリズムは時系列1細胞RNA-seqデータから転写因子ネットワークを現実的な計算時間で予測できます。擬時間に沿った転写因子の発現変化を、転写因子数分の次元の線形常微分方程式に基づきモデル化しました。制御ネットワークは、転写因子数×転写因子数の行列で表します。この行列を効率的に最適化するため、低次元の微分方程式を構築し、低次元の微分方程式を線形回帰と行列変換により元の転写因子のモデルを学習する理論を構築しました。SCODEは、精度評価において、ほかの手法と比較して、おおよそ最良の結果を示し、実行時間も非常に小さいことを示しました。
 MeSH ORA framework: MeSHによる遺伝子セット解析ツール用R/Bioconductorパッケージ
MeSH ORA framework: MeSHによる遺伝子セット解析ツール用R/Bioconductorパッケージ
MeSHは、Gene Ontologyではカバーできない多くの医学・薬学などの情報を持つ用語集で、文献の整理に使われます。我々はMeSHを遺伝子に割り当てることで、ある遺伝子セットの機能を類推する方法を提案しました。この遺伝子セット解析が実行できるR/Bioconductorパッケージを実装し、公開しました。
 Bayes Linux and Cloud Computing: バイオインフォマティクス環境をクラウド上の仮想スーパーコンピュータに展開できるソフトウェア群
Bayes Linux and Cloud Computing: バイオインフォマティクス環境をクラウド上の仮想スーパーコンピュータに展開できるソフトウェア群
たくさんのソフトウェアとデータベースを組み合わせて、複雑な計算を行うバイオインフォマティクス解析環境を自力で構築するのは困難です。そのような解析を実行するためには、適切な計算機とソフトウェア環境が必要になります。このような解析環境をクラウド上の仮想的なスーパーコンピューターに構築し、すぐに研究開発、データ解析が実施できるようBayes Linuxを開発しました。これらの環境は、ソフトウェアによって自動的にソフト・ハードウェア構築するDevOps技術を駆使して開発されています。